Che cosa [diavolo] è ENCODE
 La prima volta che ho sentito parlare di ENCODE ero entusiasta: “Una Enciclopedia degli elementi del DNA nello stesso periodo in cui la storica enciclopedia in carta Britannica chiude i battenti. Tempismo perfetto!”. Curioso e nerd, ho voluto investigare meglio la scoperta, ma non è stato facile: sebbene si tratti sempre di biologia, bisogna avere un bel bagaglio informatico e di matematica per leggere e comprendere nel dettaglio i 30 e passa articoli pubblicati simultaneamente su Nature e Genome Research.
La prima volta che ho sentito parlare di ENCODE ero entusiasta: “Una Enciclopedia degli elementi del DNA nello stesso periodo in cui la storica enciclopedia in carta Britannica chiude i battenti. Tempismo perfetto!”. Curioso e nerd, ho voluto investigare meglio la scoperta, ma non è stato facile: sebbene si tratti sempre di biologia, bisogna avere un bel bagaglio informatico e di matematica per leggere e comprendere nel dettaglio i 30 e passa articoli pubblicati simultaneamente su Nature e Genome Research.
I ricercatori hanno fatto un eccellente lavoro di divulgazione scientifica, a mio avviso, ed hanno ben curato la parte comunicativa del progetto. In particolare, la rivista Nature fornisce una guida interattiva ai documenti pubblicati per facilitare le conclusioni sul progetto. Occorre molto tempo per attraversare questa marea di informazioni, e ho quindi preferito informami attraverso del giornalismo scientifico, che avrebbe digerito le parti più complesse della notizia e provveduto a servire un facile riassundo pre-digerito. Ma qui è arrivato il secondo problema: i media riportano solo i risultati della ricerca e non il principio logico o i veri e propri esperimenti fatti per arrivare a conclusioni del tipo: “l’80% del nostro codice genetico è funzionale”.
Non contento, e per capire che cosa (diavolo) fosse ENCODE, ho incontrato Carlos Talavera, bioinformatico e dottorando al Karolinska Institutet, attualmente impegnato nell’ultimo sforzo biotecnologico SciLife, dipartimento ai confini del campus universitario che lavora su “omico”. In passato Carlos ha lavorato con uno dei gruppi di ricerca direttamente coinvolto nel consorzio, concentrandosi sull’estrazione di [tutto] l’RNA cellulare, passaggio essenziale per alcuni esperimenti di ENCODE. Un’altro vantaggio è che Carlos era inizialmente un biologo ed un wet-lab (scienziato che lavora con provette e cellule) prima di passare a fare ricerca dietro la scrivania, quindi comprende la mia “confusione sull’argomento”.
Che cos’é ENCODE?
ENCODE è un consorzio di scienziati da tutto il mondo lanciato dalla US National Human Genome Research Institut e supportato da numerosi altri instituti di ricerca. Il gruppo ha uno scopo semplice: dare un significato (ovvero trovare la funzione) alle 3 bilioni di lettere che compongono il nostro corredo genetico.
Perché c’é stato il bisogno di un progetto del genere?
Fino a qualche anno fa, è possibile che qualche professore di biologia delle scuole superiori dicesse che più del 95% del genoma* umano fosse spazzatura. (*in questo contesto, genoma=patrimonio genetico). Infatti, quando il progetto genoma umano cominciò a produrre i primi risultati, dimostrò che le nostre cellule contengono le informazioni sufficienti per circa 20.000 geni. Da qui la domanda: non c’é davvero bisogno di 10 alla 9 paia lettere per codificare così “poche” proteine? In proporzione, dei circa 2 metri di DNA che ogni singola cellula contiene, solo 4.5 cm sono usati per produrre proteine. La domanda è: ed i rimanenti 1 metro e 95cm? Negli anni successivi abbiamo capito che il DNA ha altri ruoli oltre a quello di contenere le informazioni per codificare proteine: la sua forma, le sue modifiche chimiche ed il modo in cui è organizzato nello spazio sono tutti elementi che, nel loro insieme, determinano l’identità di una specifica cellula, ed in generale l’identità di un topo rispetto a quello di una mucca o di una pianta.
Perché l’ipotesi del DNA-spazzatura ha preso piede?
Qualcuno ha suggerito che il DNA che non codifica per proteine (non coding-DNA-sequence o nCDS) doveva essere il risultato di scarto dell’evoluzione: pezzi non necessari che sono rimasi intrappolati nelle cellule di tutti i mammiferi e di cui non ci siamo ancora liberati. In linea di principio, zone del DNA che non servono dovrebbero essere perse con il passare del tempo, seguendo gli stessi principi della selezione naturale descritta da Darwin.
Cosa hanno fatto i ricercatori di ENCODE per provare che quel 1.95 metri di DNA che non codifica proteine, in qualche modo, hanno una loro funzione?
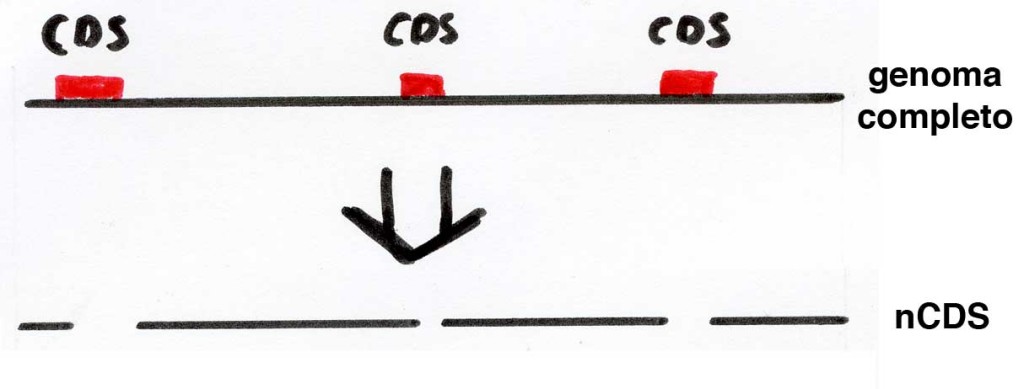
La risposta l’ha portata la bioinformatica! Gli scienziati hanno usato sofisticati programmi per processare l’intero genoma umano, lo stesso pubblicato più di 10 anni fa. Come primo passo, si sono liberati di tutte le parti che già sappiamo avere una funzione: il DNA che codifica per proteine, o CDS.
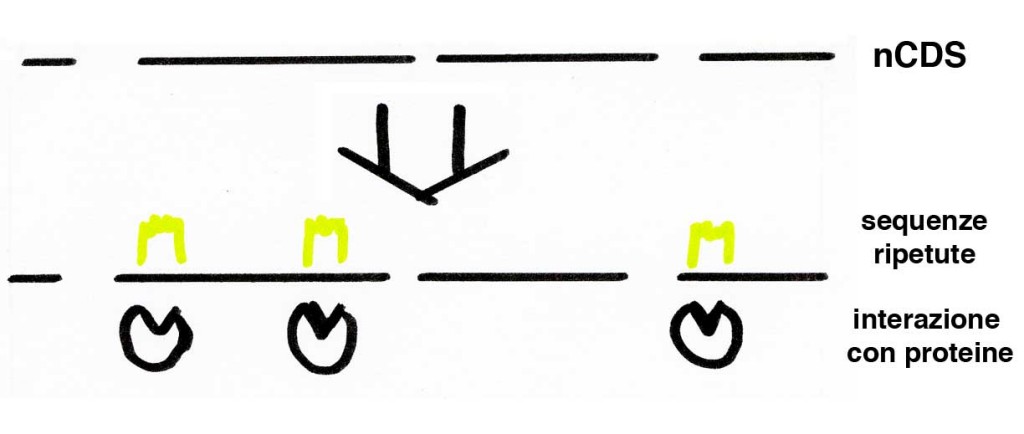
Con la parte rimanente, gli scienziati hanno usato un programma chiamato Hidden Markov Model (HMM) che individua sequenze che si ripetono più volte in una lunga stringa di dati. Il principio è lo stesso dei programmi di riconoscimento vocale: quei programmi registrano la tua voce e cercano di trovare elementi che si ripetono, come la lettera E nella parola “elettrone”, per associarvi un significato (ad esempio che quel suono significhi E). Per il DNA, il programma HMM usa la stessa logica per identificare delle zone di DNA che si ripetono, magari una a 54 centimetri e un’altra al metro 1.63 e 1.95. La logica dietro a questo esperimento è che se queste sequenze sono lì e sono ripetute, devono essersi conservate attraverso la selezione naturale, e quindi devono avere una funzione. Se così non fosse, le zone di DNA nCDS sarebbero lettere disposte a caso.
È bastato questo a convincere la comunità scientifica che nCDS ha una funzione? Il fatto che non contenga lettere messe a caso?
Assolutamente no. La logica di questo ragionamento non è esattamente impeccabile in senso biologico. Ci sono numerosissimi esempi in cui sequenze genetiche molto diverse tra loro hanno la stessa funzione, soprattutto in microrganismi come batteri e virus. Anche per questo motivo gli scienzitai hanno deciso di validare le loro “sequenze conservate”, verificando che queste zone del DNA venissero in contatto diretto con anche solo una delle 20.000 proteine della cellula. E cosí sembra essere! Zone abbandonate di DNA continuano ad avere specifiche interazioni con delle molecole, quindi, si legge nell’articolo, hanno una funzione.
È bastato questo per coprire l’80% del genoma?
No, c’é dell’altro. I ricercatori hanno quindi escluso queste nuove zone, e sono rimasti con altro materiale non ‘conservato’, che non codifichi nessuna proteina e che non interagisca con nulla. Eppure, i ricercatori di ENCODE hanno trovato pezzi di RNA (la copia carbone del DNA, essenziale per trasmetterne il messaggio) che appartengono a zone ancora inesplorate. Trovare copie di RNA in una zona apparentemente deserta del DNA è come trovare acqua su Marte. È segno che quella zona non è inabitata: ogni tanto di li passa una RNA polimerasi, l’enzima che trascrive le informazioni del DNA. Conclusione? Anche quei segmenti di DNA hanno una ‘funzione’: sono effettivamente trascritti.
Alla fine della nostra chiacchierata, Carlos mi informa che questi sono solo i dati preliminari di ENCODE. In programma abbiamo un secondo appuntamento per continuare ad esplorare uno dei più discussi ed affascinanti esperimenti della biologia degli ultimi 10 anni, di cui voglio sapere ancora di più.
…to be continued

![Che cosa [diavolo] è ENCODE](http://www.prometeusmagazine.org/wordpress/wp-content/uploads/et_temp/rollinggrande-01-7430_186x180.png)